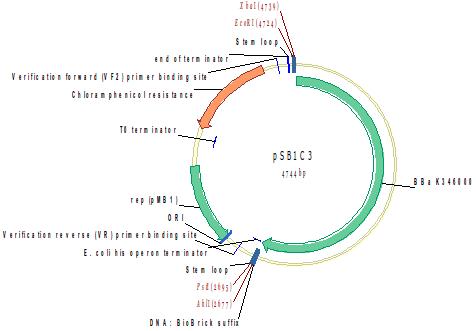
RBS(B0032)+T3 DNA-зависимая RNA-полимераза
RNA полимераза бактериофага T3 является DNA-зависимой RNA-полимеразой с высокой афинностью и специфичностью по отношению к матрице. Как и T7 полимераза, обалает довольно простой структурой по сравнению с бактериальной RNA-полимеразой, в связи с этим может широко использоваться в синтетической биологии.
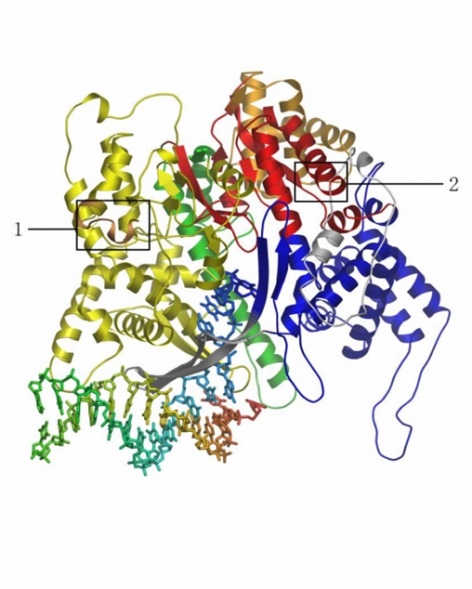
Рисунок 1 Предсказанная структура, основанная на данных для T7 полимеразы.
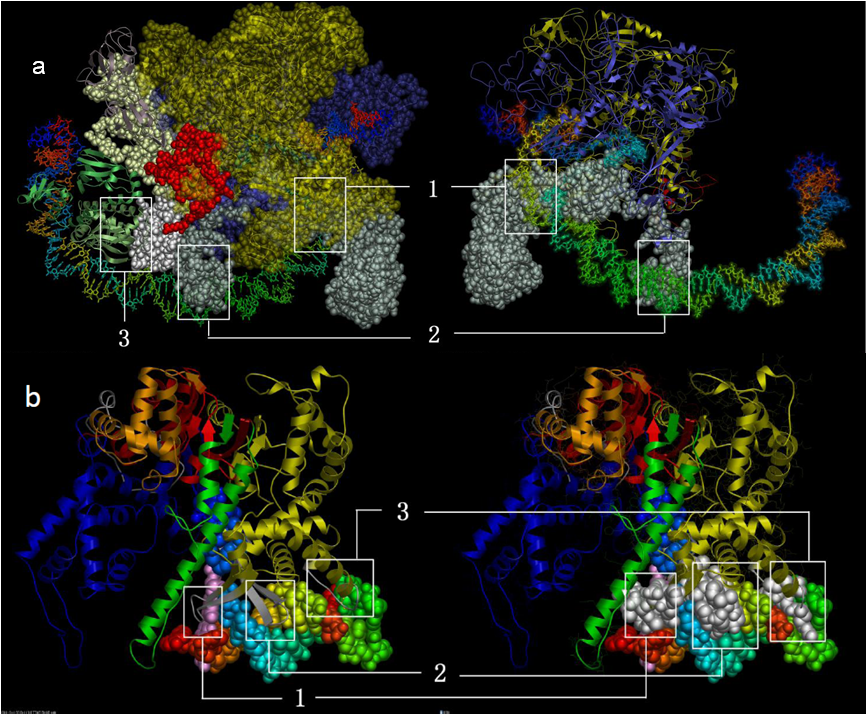
Figure 2. Предсказанное взаимодействие между T3 полимеразой и T3 промотором, основанное на данных для T7 полимеразы.
- a. Взаимодействие между бактериальной RNA полимеразой и DNA промотором.
- 1. -10 регион взаимодействует с sigma фактором;
- 2. -35 регион взаимодействует с sigma фактором;
- 3. Аlpha CTD контактирует с белком CRP.
- b. Взаимодействие между T3 полимеразой и T3 промотором.
- 1. -1 -4 TATA взаимодействует с интеркалирующей шпилькой 228 - 245.
- 2. -12 -17 регион промотора взаимодействует с петлей T3 RNA-полимеразы.
- 3. -5 -12 регион промотора взаимодействует с core specificity determinant.
Стоит отметить, что T3 RNA-полимераза обладает очень высокой аффиностью к матрице, и даже небольщая «протечка» её экспрессии может сильно увеличить базальный уровень T3 промотора. В связи с этим, её экспрессию необходимо жестко регулировать.
Литература
- Davis, R. W. & Hyman, R. W (1971)J. Mol. Biol 62, 287-301.
- McGraw, N. J., Bailey, J. N., Cleaves, G. R., Dembinski, D. R., Gocke, C. R., Joliffe, L. K., MacWright, R. S.&McAllister, W. T. (1985) Nucleic Acids Res. 13, 6753–6766.
- Klement, J. F., Moorefield, M. B., Jorgensen, E. D., Brown, J. E., Risman, S. & McAllister, W. T. (1990) J. Mol. Biol. 215, 21–29.
- Lee, S. S. & Kang, C. W. (1992) Biochem. Int. 26, 1–5.
- Li, T., Ho, H. H., Maslak, M., Schick, C. & Martin, C. T. (1996) Biochemistry 35, 3722–3727.
- Chapman, K. A. & Burgess, R. R. (1987) Nucleic Acids Res. 15, 5413–5432.
Источник: http://parts.igem.org/Part:BBa_K346000
Разработчик: Haoqian Zhang and Weiye Wang Группа: iGEM10_Peking (2010-09-25)
Выделение плазмидной ДНК
Проводилась трансформация клеток E. coli, штамм Nova-Blue (Novagen). Снятую с агара бактериальную колонию выращивали в среде LB (200 мл) в течении 18 часов. Выделение плазмидной ДНК проводили щелочным лизисом. Дополнительно обрабатывали полученную плазмидную ДНК РНКазой, проводили фенол-хлороформную экстракцию и осаждение этанолом.
Определение концентрации
Методом горизонтального электрофореза (рисунок 4-1, дор.1). Нанесение 1 мкл плазмидной ДНК. Длина линейной формы плазмиды 4744 п.н.
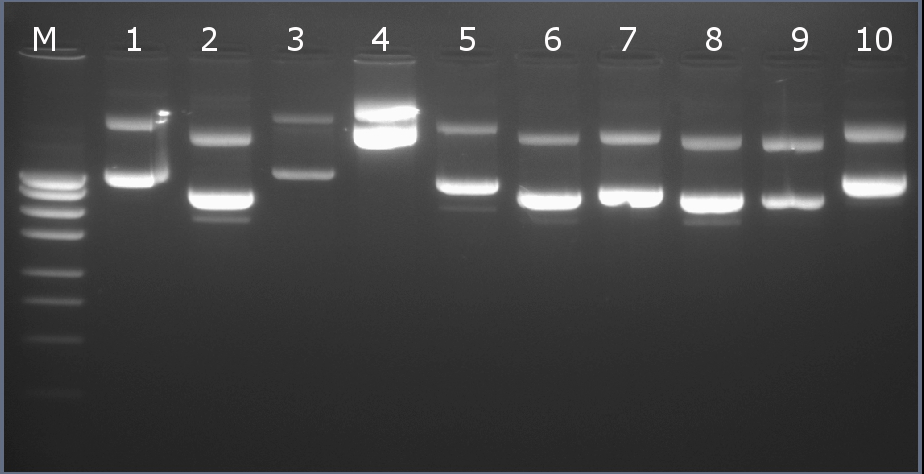
Рисунок 4-1. Электрофореграмма плазмидной ДНК. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).
Рестрикционное картирование
Рестрикционное картирование проводили с помощью эндонуклеаз рестрикции EcoRI, PstI. Разделение продуктов реакции проводили методом горизонтального электрофореза в 1,5% агарозном геле (рисунок 4-2, дорожка 1).Ожидаемая длина фрагментов 2029 п.н., 2715 п.н.
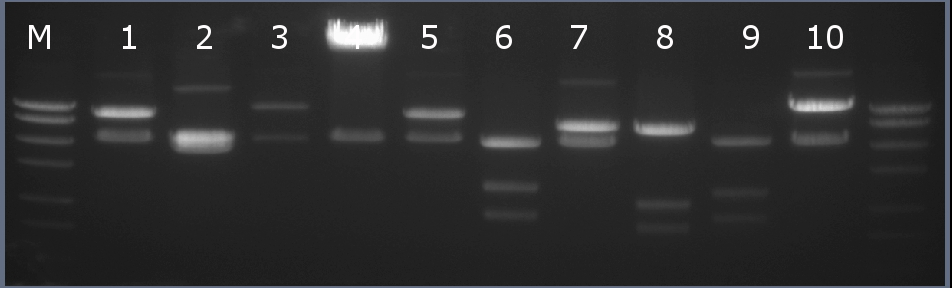
Рисунок 4-2. Электрофореграмма продуктов рестрикции. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).