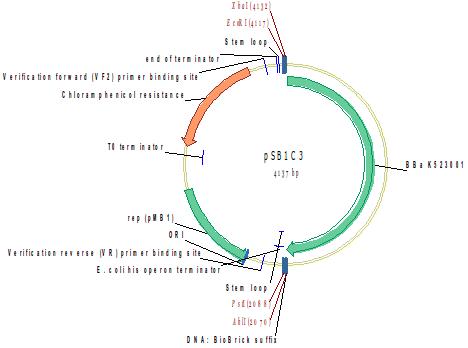
RBS + malS (периплазматическая α-амилаза E. coli)
Ген malS, кодирующий периплазматическую α-амилазу E. coli (see info from GenBank: U00096.2) с нативным сайтом связывания рибосом.
Применение в биологии
Считается, что данный белок способен разлагать крахмал, проникающий в периплазматическое пространство. В природе его основная функция в E. coli – деградировать более короткие глюкозные цепи. Lengsfeld et al (2008): "MalS продуцирует в основном мальтогексаозу из более длинных мальтодекстринов в периплазме".
Больше полезных фактов о белке MalS в публикации Spiess et al (1997).
Источник: http://parts.igem.org/Part:BBa_K523001
Разработчик: Mun Ching Lee, Sylvia Ispasanie, Allan Crossman Группа: iGEM11_Edinburgh (2011-07-19)
Выделение плазмидной ДНК
Проводилась трансформация клеток E. coli, штамм Nova-Blue (Novagen). Снятую с агара бактериальную колонию выращивали в среде LB (200 мл) в течении 18 часов. Выделение плазмидной ДНК проводили щелочным лизисом. Дополнительно обрабатывали полученную плазмидную ДНК РНКазой, проводили фенол-хлороформную экстракцию и осаждение этанолом.
Определение концентрации
Методом горизонтального электрофореза (рисунок 4-1, дор.6). Нанесение 1 мкл плазмидной ДНК. Длина линейной формы плазмиды 4137 п.н.
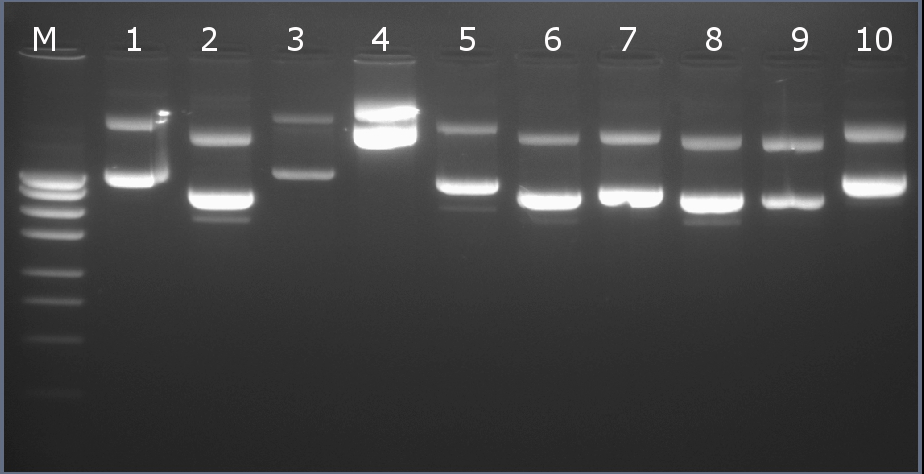
Рисунок 4-1. Электрофореграмма плазмидной ДНК. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).
Рестрикционное картирование
Рестрикционное картирование проводили с помощью эндонуклеаз рестрикции EcoRI, EcoRV. Разделение продуктов реакции проводили методом горизонтального электрофореза в 1,5% агарозном геле (рисунок 4-2, дорожка 6).Ожидаемая длина фрагментов 1969 п.н., 1269 п.н., 896 п.н., 63 п.н.
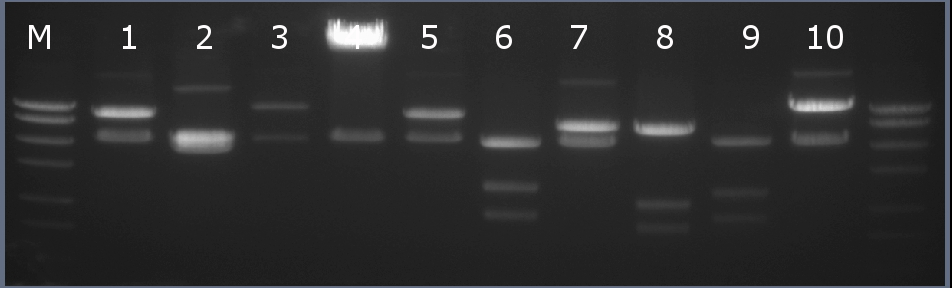
Рисунок 4-2. Электрофореграмма продуктов рестрикции. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).