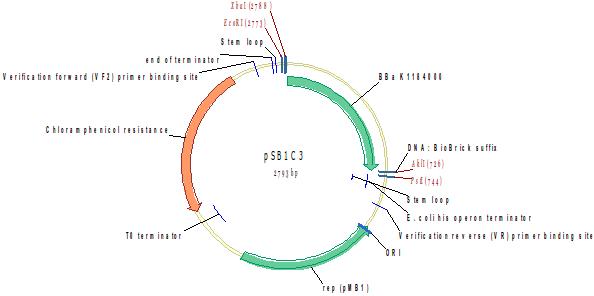
Красный киллер (KillerRed)
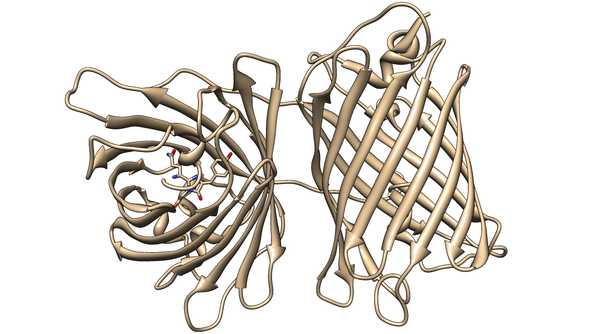
Кристаллическая структура KillerRed (PDB ID: 3GB3) с QYG хромофором
KillerRed – красный флюоресцентный белок, генерирующий активные формы кислорода (ROS) в присутствии желто-оранжевого света (540-585 nm), реагируя с водой. Разработан на основе anm2CP. Супероксиды реагируют с хромофором белка, в результате чего он темнеет. В конечном итоге происходит его обесцечивание. KillerRed по спектру сходен с mRFP1 и обладает такой же яркостью. Является олигомером и может формировать большие аггрегаты в клетках.
Последовательность оптимизирована по кодонам для клеток млекопитающих и содержит 13 редких пролиновых кодонов для E. coli (CCC) и один редкий аргининовый кодон (AGA). Тем не менее, следует принять оптимизацию во внимание, если необходимо наработать очень большие количества белка. Экспрессия в высококопийных плазмидах позволила получить детектируемый флуоресцентный сигнал.
Источник: http://parts.igem.org/Part:BBa_K1184000
Разработчик: Eric Pederson Группа: iGEM13_Carnegie_Mellon (2013-09-04)
Выделение плазмидной ДНК
Проводилась трансформация клеток E. coli, штамм Nova-Blue (Novagen). Снятую с агара бактериальную колонию выращивали в среде LB (200 мл) в течении 18 часов. Выделение плазмидной ДНК проводили щелочным лизисом. Дополнительно обрабатывали полученную плазмидную ДНК РНКазой, проводили фенол-хлороформную экстракцию и осаждение этанолом.
Определение концентрации
Методом горизонтального электрофореза (рисунок 6-1, дор. 9). Нанесение 1 мкл плазмидной ДНК. Длина линейной формы плазмиды 2793 п.н.
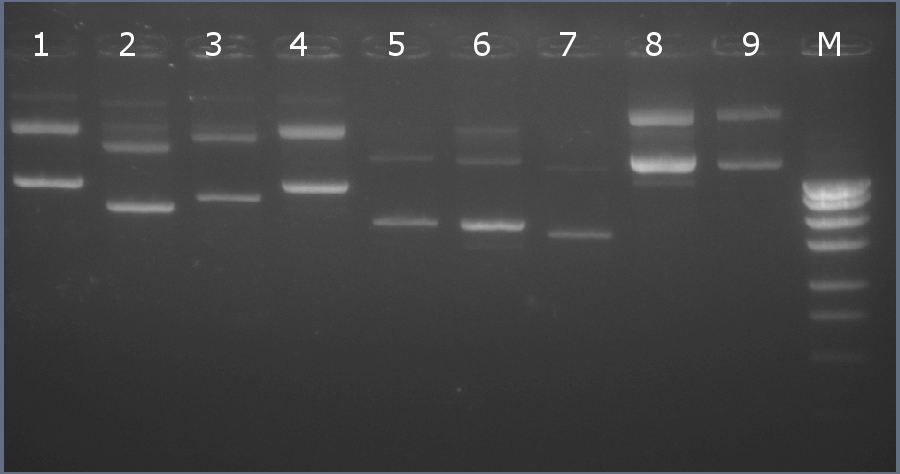
Рисунок 6-1. Электрофореграмма плазмидной ДНК. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).
Рестрикционное картирование
Рестрикционное картирование проводили с помощью эндонуклеаз рестрикции EcoRI, PstI, EcoRV. Разделение продуктов реакции проводили методом горизонтального электрофореза в 1,5% агарозном геле (рисунок 6-2, дорожка 9).Ожидаемая длина фрагментов 2029 п.н., 762 п.н.
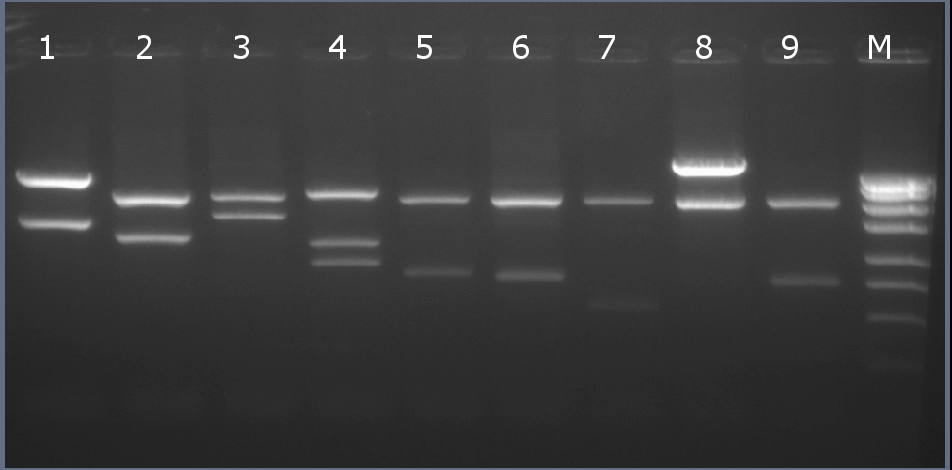
Рисунок 6-2. Электрофореграмма продуктов рестрикции. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).