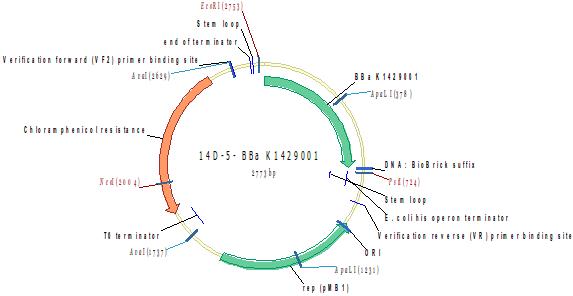
Кодирующий регион красного флюорецентного белка (Red Fluorescent Protein (RFP)
Синетический синий флюоресцентный белок с сайтом связывания рибосом. Происхождение не связано с Aequorea.
Применение в биологии
Может быть использован как репортер. Продукция RFP в E. coli под контролем промотора T7 обозначена ниже как "RFP2" (фрагмент клонирован в pUC19):

Спектры возбуждения и излучения соответствуют спецификациям на сайте DNA2.0.
Источник: http://parts.igem.org/Part:BBa_K1429001
Разработчик: Ramsez McCall Группа: iGEM14_Genspace (2014-09-23)
Выделение плазмидной ДНК
Проводилась трансформация клеток E. coli, штамм Nova-Blue (Novagen). Снятую с агара бактериальную колонию выращивали в среде LB (200 мл) в течении 18 часов. Выделение плазмидной ДНК проводили щелочным лизисом. Дополнительно обрабатывали полученную плазмидную ДНК РНКазой, проводили фенол-хлороформную экстракцию и осаждение этанолом.
Определение концентрации
Методом горизонтального электрофореза (рисунок 2-1, дор.6). Нанесение 1 мкл плазмидной ДНК. Длина линейной формы плазмиды 2773 п.н.
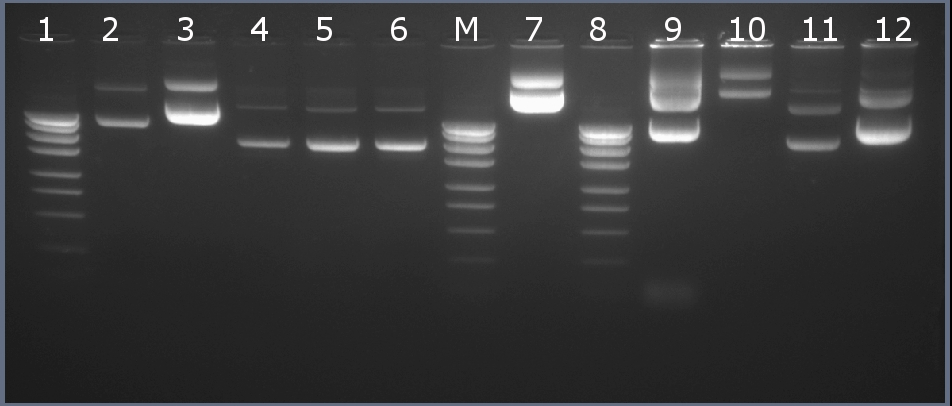
Рисунок 2-1. Электрофореграмма плазмидной ДНК. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).
Рестрикционное картирование
Рестрикционное картирование проводили с помощью эндонуклеаз рестрикции EcoRI, PstI. Разделение продуктов реакции проводили методом горизонтального электрофореза в 1,5% агарозном геле (рисунок 2-2, дорожка 6).Ожидаемая длина фрагментов 2029 п.н., 744 п.н.
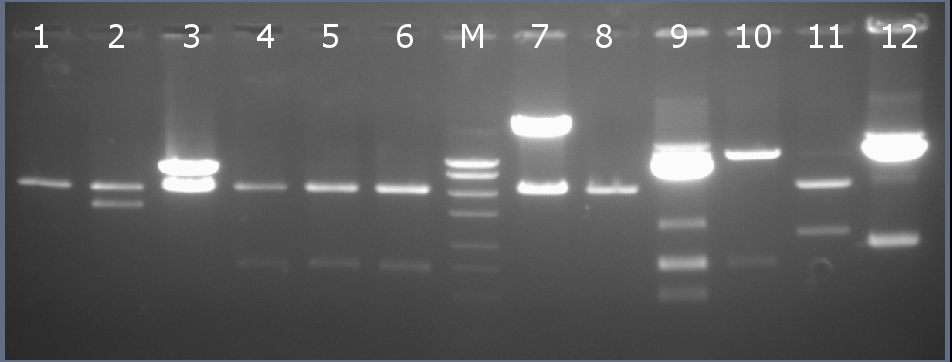
Рисунок 2-2. Электрофореграмма продуктов рестрикции. М – маркерная лестница p250 (250, 500, 750, 1000, 1500, 2000, 2500, 3000 п.н.).